Gímszarvas csoport

Magyar kutatók megfejtették a csodaszarvas géntérképét,
meghatározták a gímszarvas teljes genomi DNS szekvenciáját
Kulturális áttekintés
A
gímszarvas teljes genomi DNS szekvencia meghatározási ambíciót nem kis
mértékben motiválta az a kb. 20000 év, amely a szarvast az emberi kultúrához
köti. A szarvas emblematikus, kultikus lény, amelyet tisztelet és csodálat
övez: már jelen van a jégkorszaki barlangrajzokon, több ezer éves ékírásokban,
művészi szobrocskák őrzik alakját. Irodalmi szereplő az ókor óta, versek és
mitológiák hőse (a magyar őstörténet egyik fő alakja), zeneművek,
képzőművészeti, film és fotóművészeti alkotások, egyházi énekek őrzik alakját.
A gímszarvas, Cervus elaphus genom program, CerEla1.0, (Wonder
Deer Genome)
A Mezőgazdasági Biotechnológiai
Központ (NAIK-MBK, Gödöllő), a Kaposvári Egyetem Állattenyésztési, az ELTE
Genetikai, és a SOTE 1. Belgy. Klinika, a Szent
István Tudomány Egyetem, Szegedi Biológiai Kutatóközpont MSc
és PhD programjai összefogásával, kizárólagosan hazai erőforrásokból, elkészült
a gímszarvas DNS-ének szekvenciája és teljes genomjának leírása, amely a
CerEla1.0 nevet kapta. A CerEla1.0 a gímszarvas genom első leírása a világon.
Lényegét tekintve a Humán Genom Program gímszarvas megfelelője. A CerEla1.0
program egy hosszú út végét jelzi, amely 1998-ban indult, koncepcióját Orosz
László dolgozta ki, az állattenyésztési és vadgazdálkodási hasznosítást Horn Péter,
a klinikai irányt Lakatos Péter jegyezte. Összesen több mint 50 diák és kutató
munkatárs járult hozzá a program haladásához. A programot jelentős OTKA,
NKTH (Széchenyi Program), FVM, EÜM és MTA pályázati források támogatták az első
9-10 évben. Ezt követően nagyrészt a Kaposvári Egyetem állattenyésztési,
vadgazdálkodási és természetvédelmi doktori iskolája és a Földművelésügyi
Minisztérium tudományos gyakornoki programja biztosította a költségeket. A CerELa1.0 genom szabadon elérhető és letölthető az Egyesült Államokban gondozott
nemzetközi genom adatbázisból (NCBI, MKHE00000000 azonosítóval), továbbá
hazánkban a NAIK-MBK honlapján is megtekinthető a http://emboss.abc.hu/wonderdeer/JBrowse
elérési útvonalon.
A CerEla1.0-ről beszámoló
tudományos közlemény a Molecular Genetics
and Genomics –ban (MGG)
2018 Január 2-án jelent meg online : „Nóra Á. Bana, Anna Nyíri, János Nagy,
Krisztián Frank, Tibor Nagy, Viktor Stéger, Mátyás
Schiller, Péter Lakatos, László Sugár, Péter Horn, Endre Barta, László Orosz: The red deer Cervus elaphus
genome CerEla1.0: sequencing,
annotating, genes, and chromosomes”. Lásd a lapalján.
A „Csodaszarvas” genom: CerEla1.0 elkészítése
2011-ben egy csodálatosan szép, kapitális gímszarvasbika
vérmintájából vontuk ki a DNS-t. A legmodernebb eljárások egyikével (ILLUMINA Technology) meghatároztuk a DNS szekvenciáját (a bázisok sorrendjét), majd tovább rendeztük egy számítógép programcsomag
segítségével (ALLPATH-LG). A szarvasbika (Crot 3016, a mellékelt, legfelső fénykép közepén látható, 7
éves korában 2010-ben) a Kaposvári Egyetem Vadgazdálkodási központja telepén,
Bőszénfán természet közeli körülmények között élt. A DNS szekvencia ismeretében
változatos számítógépes eljárások alkalmazásával meghatároztuk az egyes
géneket, a gének szerkezetét, a gének szabályozó részeit és egyéb fontos
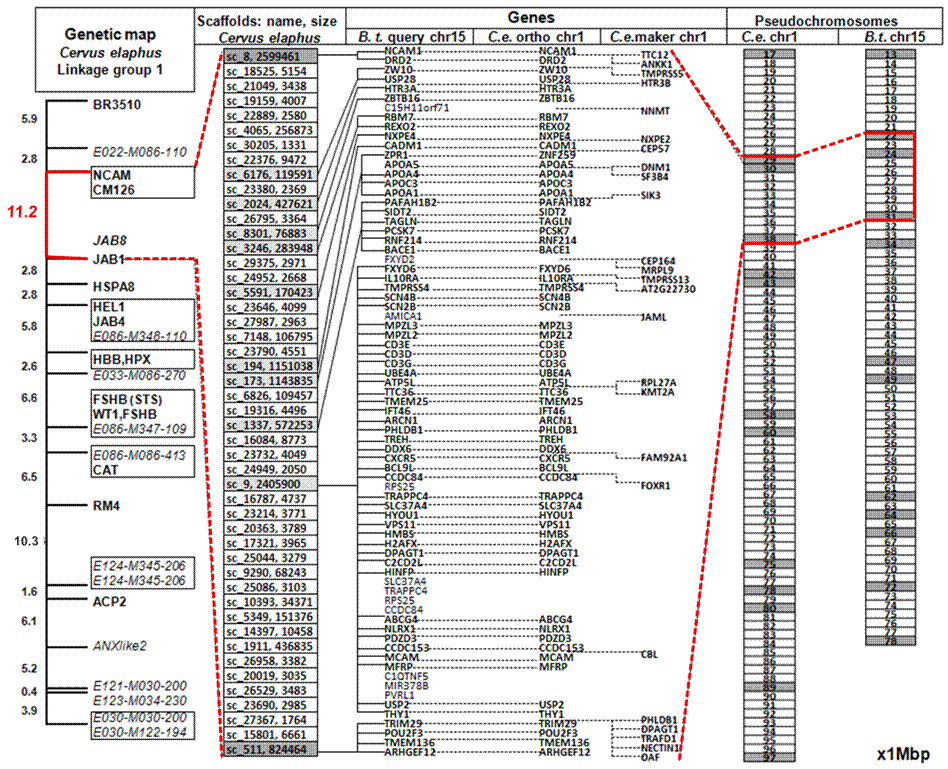
elemeket. A genetika két alaptételét alkalmazva határoztuk meg, hogy az egyes
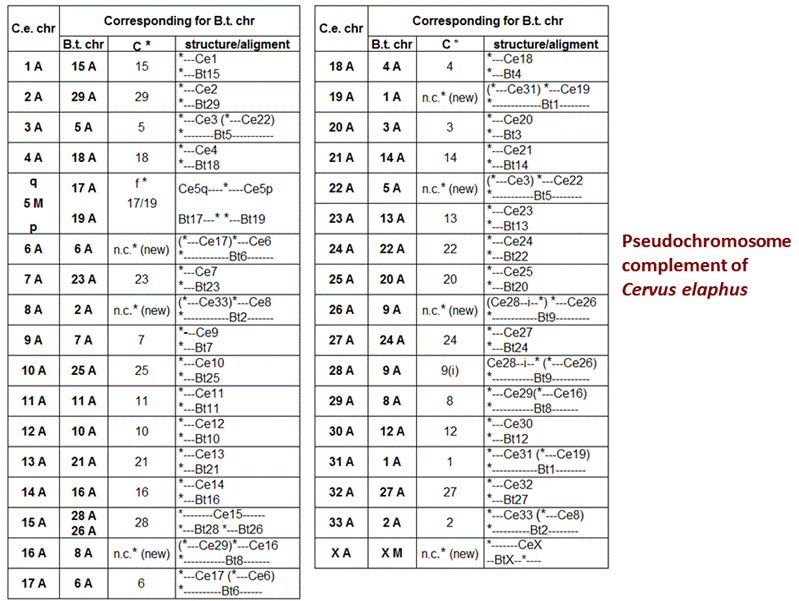
gének mely kromoszómákon helyezkednek el és milyen sorrendben. Az egyik
kiinduló pont a kromoszóma és a géntérkép ko-linearitási
tétele, a másik az összehasonlító géntérképezés elve volt. A gímszarvas
géntérkép pontjai hosszú szakaszokon azonos sorrendben találhatóak a
szarvasmarha genomban is, azaz kiterjedt szinténiák,
lokális kapcsoltságok figyelhetőek meg.
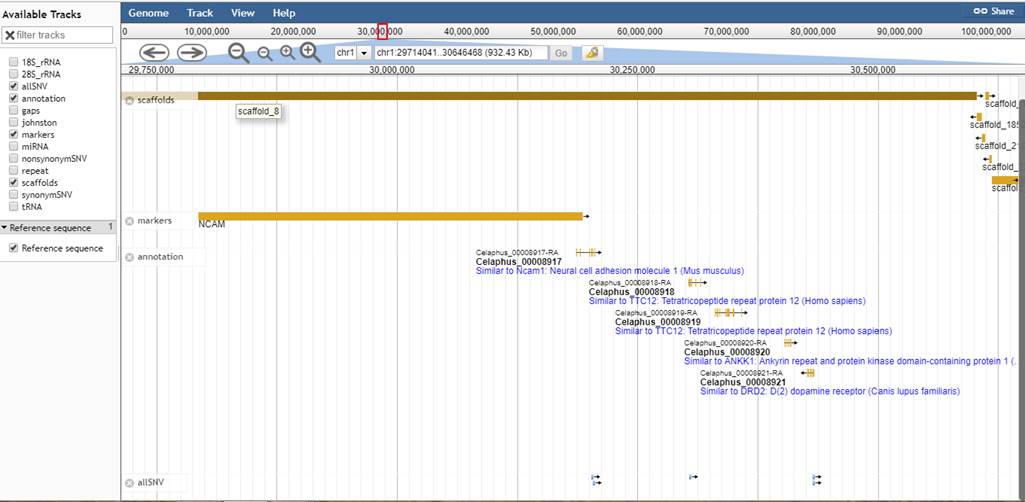
Egy további elemzésben meghatároztuk az egyes
kromoszómákon az un. centromeronok helyét. Ez
utóbbiak biztosítják sejtosztódáskor a kromoszómák precíz vándorlását az
utódsejtekbe. A gímszarvas bika genomját 33 Autoszóma DNS-e, valamint az X és az Y
kromoszóma
DNS-e alkotja (plusz a mitokondrium DNS). A 3 milliárd DNS szekvenciából 2,7
milliárd bázispárnyit sikerült meghatározni. Továbbá leírtuk a genom
legkülönbözőbb, genetikai szempontból fontos elemeit is. A fehérjét kódoló
gének közül 19368-at azonosítottunk (a kérődzők ismert génjeinek 90 %-át). A
gének részletes szerkezetét, evolúciós rokonságait, funkcióit meghatároztuk (ez
az un. „gén annotáció”). Ilyenek például a sejten, szerveken, szöveteken belüli, fejlődésben betöltött szerepek,
hasonló gének más fajokban, a gén belső felépítése (az un. exon,
intron szerkezet, 5’ és 3’ UTR-ek, stb.). Feltártuk
az ismétlődő, repetitív szekvenciákat, LTR elemeket, transzfer RNS, kis RNS és
riboszóma RNS géneket, mindezen genetikai elemek helyzetét a kromoszómák mentén
a DNS szekvenciában. A gímszarvas genom feltárása lehetőséget adott 2.8 millió heterozigóta
pont változat (SNP) és 365 ezer apró deléció (bázis
kiesés) és inszerció (bázis betoldás), az un. indelek azonosítására a genom mentén.
4. Genetikai
elemek helyzete a kromoszómák mentén.
A CerEla1.0 értékes adatokkal
bővítette a kérődzők genom/kromoszóma evolúciójáról (kariológiai
evolúció) alkotott képet is. Feltárta például, hogy milyen kromoszóma
átrendeződések történtek (kromoszómák fúziói, hasadások, inverziók, azaz egyes
szakaszok megfordulása, transzlokációk, amikor kromoszóma szakaszok
áthelyeződnek) az elmúlt 30 millió év során. Azóta, hogy a szarvasfélék (Cervidae) és a
tülkös szarvúak (Bovidae)
elszakadtak a közös kérődző „Pecora”ősüktől. Megjegyezném, hogy a mai szibériai
pézsmaszarvas áll a legközelebb ehhez az ősi állapothoz.
A CerEla1.0 felhasználásai
A
CerEla1.0-t folyamatosan bővíthető az új ismeretek függvényében, bár már most isgazdag forrása a populációelemzéseknek, a leszármazási és
az eredet vizsgálatoknak, a forenzikus, a bűnügyi, a
régészeti, a természetvédelmi és a vadgazdálkodási felhasználásoknak. Teljes
genom asszociációs vizsgálatokra (GWAS, Genome Wide Assotiation Studies) ad
lehetőséget. Egy ilyen GWAS vizsgálat lehet a jövőben például az aranyérmes
agancsok titkának megfejtése is. Az utóbbi években a CerEla1.0 számos
természetvédelmi célú genom kutatás sikeres elindításának jelentett és jelent
bátorítást a „gímszarvas genomon” felnőtt fiatal kutatóknak, ilyen például a
nagy ragadozók (medve, farkas, hiúz) magyarországi
előfordulásának/vándorlásának igazolása, a vaddisznó állományok nyomon követése
genom diagnosztikával, vagy a pannon méh genomjának feltárása és a DNS
vizsgálat méhészeti alkalmazása. A CerEla1.0 hasznosult már eddig is, és hasznosulhat még a
jövőben is egyes orvosi biológiai kutatásokban (pl. oszteoporózis,
szervfejlődés/regeneráció, robusztus szövet gyarapodás/tumor biológia).
Vadászaszati Világkiállítás 2021 és CerEla1.0
A
Vadászati Világkiállítást 2021-ben Magyarország rendezi. Vadászati/trófea
minőségét tekintve Magyarországon, elsősorban Gemencen és a Dél Dunántúlon él a
világ legkiválóbb gímszarvas populációja. Nemzeti kincs. A gímszarvas programot
megvalósítók örömére lenne, ha a Világkiállítás Magyar Pavilonjában a rekord
trófeák mellett a CerEla1.0 is helyet kapna.
Kapcsolódó cikkek
|
Molecular Genetics
and Genomics The red
deer Cervus elaphus genome CerEla1.0: sequencing, annotating, genes, and chromosomes |
|